

数据降维聚类工具介绍——SpatialPCA
描述
现有常用的空间转录组降维聚类方法,如主成分分析(principal component analysis,PCA)、非负矩阵分解(non-negative matrix factorization,NMF)等,多是基于单细胞数据进行开发的,因此不一定完全适用于空间转录组数据。例如,常用的降维聚类方法并没有运用空间转录组所特有的组织空间定位信息,而仅仅只是基于表达谱进行聚类。但是,对于生物组织,相邻的位置通常都具有相似的细胞组成和相似的基因表达水平。因此,将空间位置信息纳入到降维聚类过程中,将更有利于实现接近生物实际状态的空间聚类。
SpatialPCA就是基于以上观点所开发的空间转录组降维聚类工具。该工具使用所谓的空间概率PCA(spatial probabilistic PCA),能够明确地模拟组织位置间的空间相关性结构,在降维后的数据中保留原始空间数据的相邻近似性。SpatialPCA将空间定位信息作为额外的输入,使用核矩阵模拟组织位置间的空间结构。基于该方法所得到降维主成分因为包含相关的空间结构信息,称其为空间主成分。具体来说,该方法同时使用基因表达矩阵和位置信息矩阵,将基因表达矩阵构建为一个潜在因子的函数模型。同时,根据位置信息矩阵对潜在因子构建核矩阵,以明确潜在因子的空间结构相关信息(图1)。
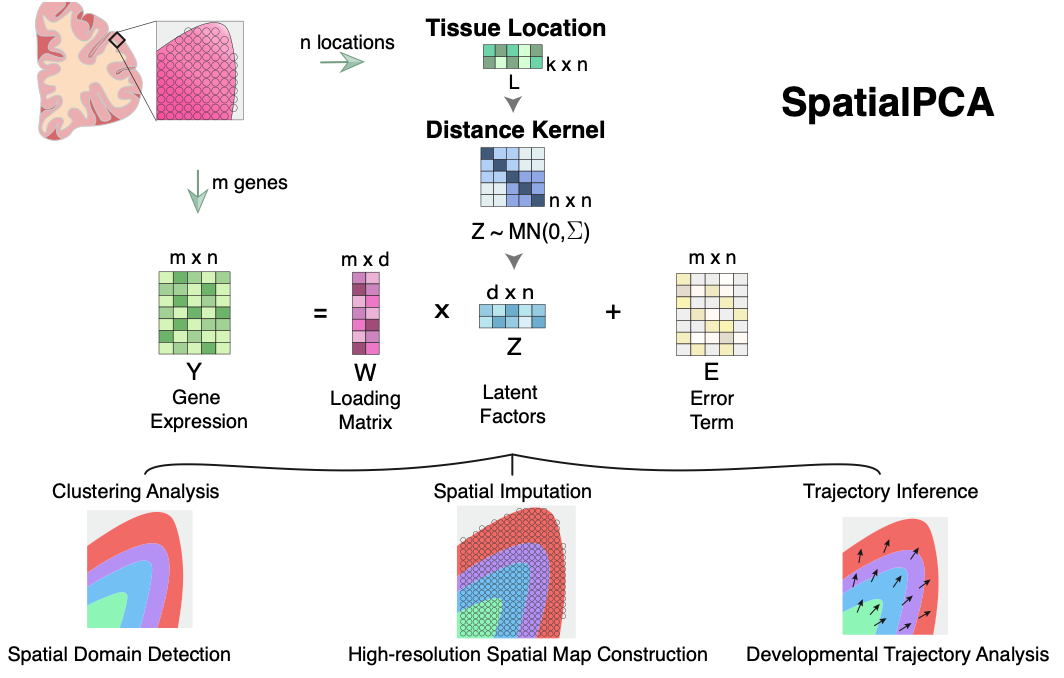
图1
这里选取了背外侧前额叶皮层(DLPFC)作为模拟数据的来源,通过手动分割各皮层,确定10000个特定细胞类型的细胞空间位置,并得到这些细胞的单细胞表达数据,设计了四种不同细胞组成情况,人工构建了四种空间转录组数据。相较于其他现有的空间聚类方法(包括采纳空间信息与不采纳空间信息的),SpatialPCA在模拟数据测试中体现出了更佳的检测性能(图2)。
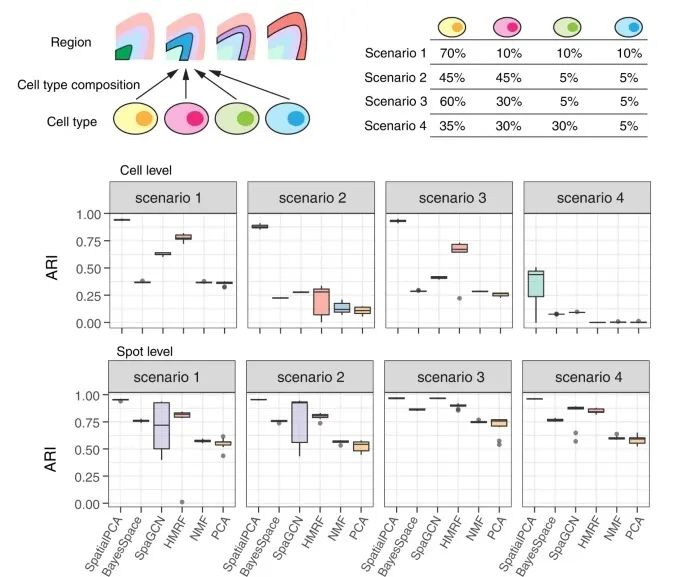
图2
在真实数据的测试中,首先选取了人类DLPFC的Visium空间转录组数据,对多种工具(SpatialPCA, BayesSpace, SpaGCN, HMRF, stLearn, PCA, NMF)进行了平行测试。结果显示,SpatialPCA生成空间聚类结果最接近实际的生物结构(图3A),其预测的精确性和预测的空间域结构连续性(CHAOS打分越低连续性越高)也是几种工具中最高的(图3B)。
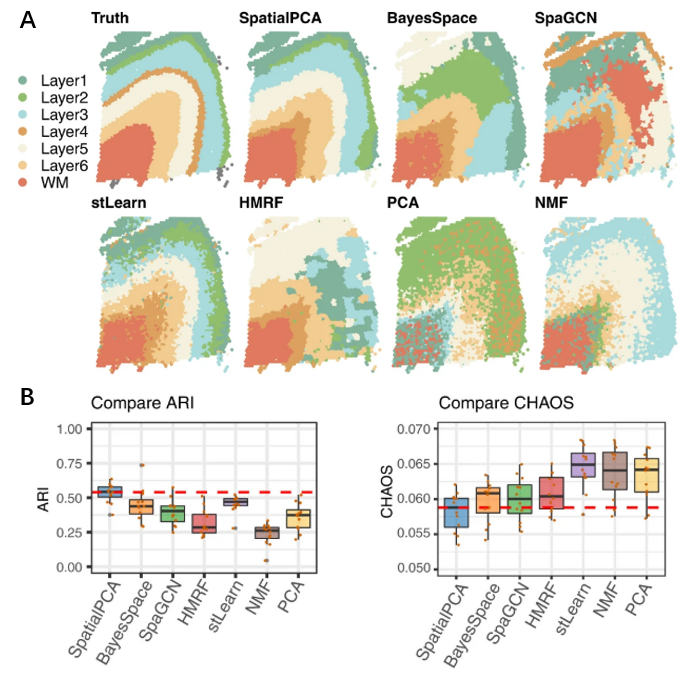
图3
为了确认SpatialPCA的技术普适性,研究人员同时还在其他技术(Slide-seq,Slide-seqV2)产生的小鼠脑组织空间转录组数据中进行了测试。结果均显示在多种工具中,SpatialPCA的聚类结果与实际情况更为符合(图4)。而为了确认SpatialPCA的样本普适性,利用HER2阳性乳腺肿瘤样本进行测试,结果也显示了SpatialPCA具有更好的聚类精确性和更符合实际情况。
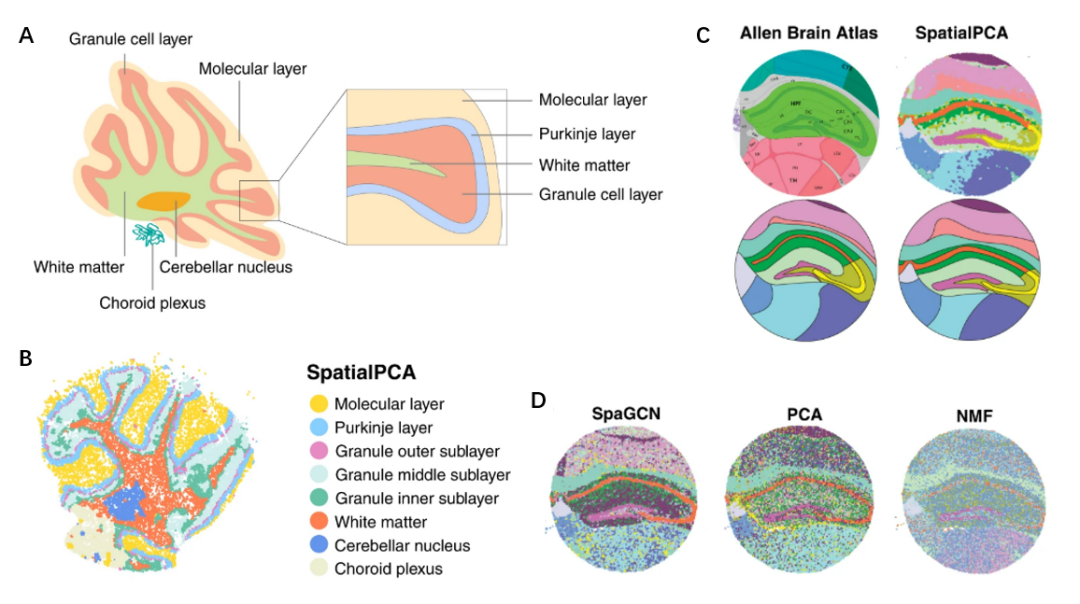
图4
由于SpatialPCA所计算出的空间主成分同时包含了空间信息和基因表达信息,因此可以据此使用一些单细胞转录组分析工具来进行下游分析。通过使用Slingshot对肿瘤组织的空间转录组数据进行了轨迹推断,显示出了一条由肿瘤区域指向肿瘤周围区域再到正常组织的轨迹(图6A),计算得到的伪时间相关基因富集于免疫反应、细胞介导免疫和吞噬识别通路,凸显了它们在癌症进展、肿瘤侵袭和转移中的重要性。
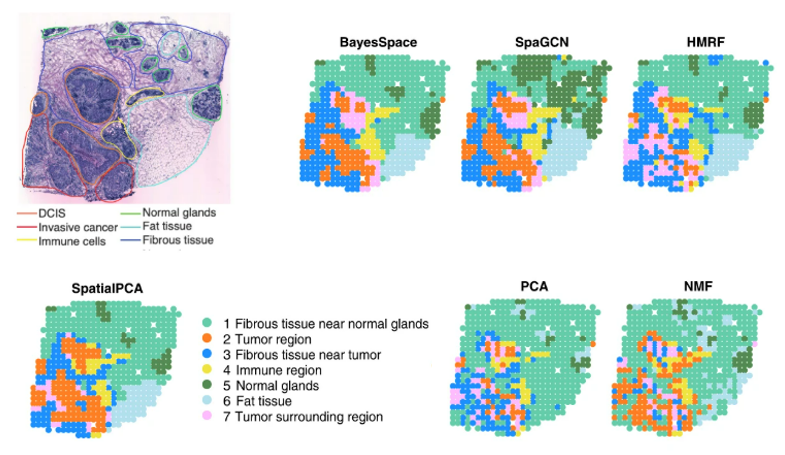
图5
另外,根据SpatialPCA的建模框架,可以在新的空间位置上推断基因表达水平,一次也可以通过低分辨率的样本数据构建高分辨率的空间图谱。对肿瘤组织的测试显示,SpatialPCA所构建的高分辨率空间图谱显示出连续与平滑的特征,其聚类精确定位了不同组织区域间的边界,并细化了紧邻肿瘤和免疫区域之外的一层薄薄的肿瘤周围区域(图6B),显示SpatialPCA在精细结构方面的优势。
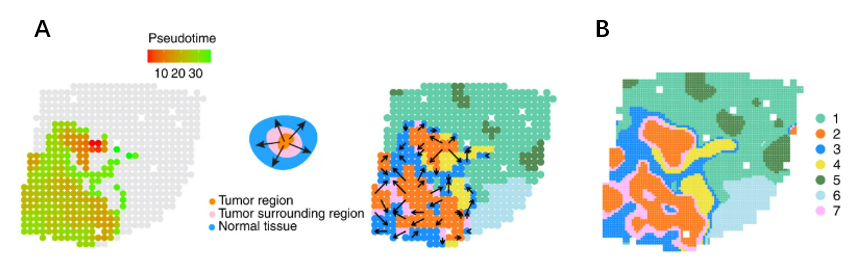
图6
SpatialPCA是一种适合针对空间转录组数据降维聚类的分析工具,如果老师们手上的空间数据分群聚类不够理想,赶快用SpatialPCA来试一试吧。
全部0条评论

快来发表一下你的评论吧 !

